Obtendo dados e plotando mapas no R - Versão 3
By Ricardo de Oliveira Perdiz in mapa R Tutorial
March 6, 2020
Continuando a reformulação de postagens antigas, reapresento uma publicada em 2016 sobre obtenção de dados e plotagem de mapas usando o R. Fiz correções no script lá apresentado, e eliminei o desnecessário (como atribuição de pasta de trabalho e caminhos relativos para ler os dados), de forma a tornar o tutorial abaixo totalmente reprodutível. Por isso, peço encarecidamente que, uma vez encontrado algum erro, favor me avisar, para que eu corrija o mais rapidamente possível e você possa fazer bom uso desta postagem.
Para reproduzir a postagem em seu computador, baixe o arquivo .Rmd juntamente com todos os dados necessários para gerar o mapa reproduzido no fim desta postagem
neste link, procure o arquivo de extensão “.Rmd” e abra-o em seu computador, preferencialmente usando o
RStudio.
Execute o arquivo usando a função Preview presente no RStudio e você terá uma página exatamente a esta aqui disponibilizada.
Certifique-se de que você tenha os pacotes knitr, rmarkdown e todas as dependências devidamente instaladas em seu computador.
Esta postagem foi construída usando o formato R Notebook. Visite os links ao fim desta postagem para obter mais informações sobre este formato e seus benefícios para a reprodutibilidade na Ciência.
O tutorial é bem simples e visa a produção de um mapa de distribuição geográfica utilizando dados que abrangem a distribuição espacial de duas espécies de Burseraceae, Protium aracouchini (Aubl.) Marchand e P. heptaphyllum (Aubl.) Marchand. A primeira planta faz parte de um complexo de espécies informalmente denominado complexo Protium aracouchini, cuja sistemática e taxonomia vem sendo objeto de estudo em meu doutorado. A segunda espécie, P. heptaphyllum, é objeto de estudo de Gabriel Damasco, colaborador do LABOTAM e aluno de doutorado na Universidade da Califórnia, Berkeley.
Os dados foram baixados do herbário virtual do Jardim Botânico de Nova Iorque que é onde trabalha o especialista em Burseraceae, Dr. Douglas Daly, e cuja coleção de espécimes e dados vêm sendo bem cuidada há algumas décadas. Para esta postagem, filtramos apenas os dados para as duas espécies citadas acima.
Carregando os pacotes
Faremos uso dos pacotes
maps (para plotar o mapa),
RColorBrewer (paleta de cores),
magrittr (pipe, ou %>%) e
dplyr (manipulação de ‘dataframes’) . É imprescindível que todos estejam atualizados e com as dependências instaladas.
library(maps)
library(RColorBrewer)
library(magrittr)
library(dplyr)
Importando os dados
Os dados estão reunidos em dois arquivos ‘.csv’ separados por tabulação e codificados em UTF-8. Baixe-os para seu computador:
aracouch_url <- "http://www.botanicaamazonica.wiki.br/labotam/lib/exe/fetch.php?media=alunos:r.perdiz:nybg_paracouchini.csv"
#protium aracouchini
aracouch <-
read.table(url(aracouch_url), header = T, as.is = T, sep = '\t', dec = '.') %>%
select(recordedBy, recordNumber, decimalLatitude, decimalLongitude, identifiedBy, specificEpithet) %>%
arrange(recordedBy, recordNumber)
aracouch
hepta_url <- "http://www.botanicaamazonica.wiki.br/labotam/lib/exe/fetch.php?media=alunos:r.perdiz:nybg_pheptaphyllum.csv"
#protium heptaphyllum
heptaphy <- read.table(url(hepta_url), header = T, as.is = T, sep = '\t', dec = '.') %>%
select(recordedBy, recordNumber, decimalLatitude, decimalLongitude, identifiedBy, specificEpithet) %>%
arrange(recordedBy, recordNumber)
heptaphy
Checa e limpa os dados
Em nosso caso, devemos verificar:
- existência de valores vazios; em caso positivo, devemos eliminá-los;
- confiabilidade dos valores de latitude e longitude, às vezes, por diversos fatores, há troca de sinais (negativos e positivos) ocasionando equívocos quanto à ocorrência exata da amostra. Se for percebido algo assim, é bom checar os dados e buscar corrigí-los.
# une os dados e passa para a proxima acao
dad <-
rbind(aracouch,heptaphy) %>%
#elimina os registros vazios de coletor
filter(recordedBy != '') %>%
#elimina os registros sem lat ou long
filter(decimalLatitude != '' | decimalLongitude != '') %>%
#filtra apenas os especimes identificados pelo especialista da família Burseraceae
filter(identifiedBy == 'D. C. Daly') %>%
arrange(recordedBy, recordNumber)
dad
Há registros duplicados! Por exemplo, a amostra A. Fernandez 4897 possui dois registros.
Devemos então buscar apenas os dados únicos.
Para isso, limparemos os dados de coletor e número de coleta para poder criar um identificador único para cada amostra e assim podermos identificar as duplicatas.
A limpeza consistirá em eliminar ‘.’, espacos vazios, apóstrofe e ‘_’ duplos das colunas recordedBy (== coletor) e recordNumber (== número de coleta).
Após a limpeza, criaremos um identificador único unindo as colunas recordedBy e recordNumber, separadas por _.
dad$recordedBy <-
gsub('\\.', '_', dad$recordedBy) %>%
gsub(' ', '_', .) %>%
gsub("'", '_', .) %>%
gsub('__', '_', .)
#faz-se o mesmo para os numeros de coleta
dad$recordNumber <-
gsub('/','_', dad$recordNumber) %>%
gsub(' ', '_', .) %>%
gsub('\\.', '_', .)
#cria o identificador de coleta e especie
dados <-
dad %>%
mutate( ID = paste(recordedBy,recordNumber, sep = '_'), Species = paste('Protium', specificEpithet, sep = ' '))
dados$ID <- gsub('__','_',dados$ID)
Nossos identificadores ficaram assim:
head(dados$ID, 100)
Reparem que as duplicatas não foram eliminadas ainda (por exemplo, C_A_Cid_Ferreira_6659).
#quem sao os dados unicos
unicos <- unique(dados$ID)
#agora filtra os dados unicos no dataframe, eliminando os duplicados
#faz-se uso da funcao match para obter esse resultado
prot <-
match(unicos, dados$ID) %>%
dados[.,]
Por fim, checamos também os dados de latitude e longitude para ver se está tudo correto.
#verifica a cobertura de lat e long para ver se estao dentro
# dos limites da America do Sul
lat <- range(prot$decimalLatitude)
long <- range(prot$decimalLongitude) #aqui tem algo estranho
#percebe-se aqui que ha valores que caem fora da Am Sul
#limite e pouco mais de -80
head(sort(prot$decimalLongitude))
#devemos eliminar
protium <-
prot %>%
filter(decimalLongitude > -80)
Cria variáveis para plotar o mapa
#vetor com epitetos especificos
spp <- unique(protium$Species)
#vetor de cores para utilizar no mapa
#para cada especie, uma cor
protium$cores.map <- ifelse(protium$Species == spp[1], 'red','black')
cores.map <- protium$cores.map
#cria um vetor de tamanho para cada especie
#como uma coluna de protium
#P heptaphyllum possui uma distribuicao mais ampla
#por isso atribuo um tamanho menor pra ela
protium$cex.p <- ifelse(protium$Species == spp[1], 1, 0.8)
# vetor contendo dados de latitude e longitude em objetos separados
lat <- protium$decimalLatitude
long <- protium$decimalLongitude
# amplitude de lat e long para lat e long
y1 <- range(lat) + c(-1,1)
x1 <- range(long) + c(-1,1)
#vetor para os simbolos pch
protium$pontos <- ifelse(protium$Species == spp[1], 21, 24)
#vetor para paises
paises <- c('Brazil','Argentina','Peru','Paraguay','Ecuador','Chile','Uruguay','French Guiana','Suriname','Venezuela','Colombia','Guyana','Bolivia','Panama','Costa Rica')
Plota o mapa
#plota o mapa
map(regions = paises, fill = F, xlim = x1, ylim = y1)
#plota os pontos de ocorrencia de cada especie
points(long,lat, pch = protium$pontos, col = cores.map, bg = cores.map, cex = protium$cex.p)
#coloca eixos das coordenadas
map.axes()
axis(side=4,las=1)
axis(side=3,las=1)
#coloca escala do mapa
par(cex=1, las=1)
map.scale(max(long) - 12, ratio = F, cex = 1, metric = T)
#plota a linha do equador
abline(h=0,lwd=0.5,lty="dotted")
#nomeia a linha do equador
text(x = max(long) - 1, y = 1,labels="Equador", cex=1, adj=c(1,0.5))
#plota uma legenda
legend(max(long) - 16 , max(lat),legend = spp, pch = unique(protium$pontos), pt.bg = unique(cores.map), cex = 0.8, x.intersp = 0.8, text.font = 3)
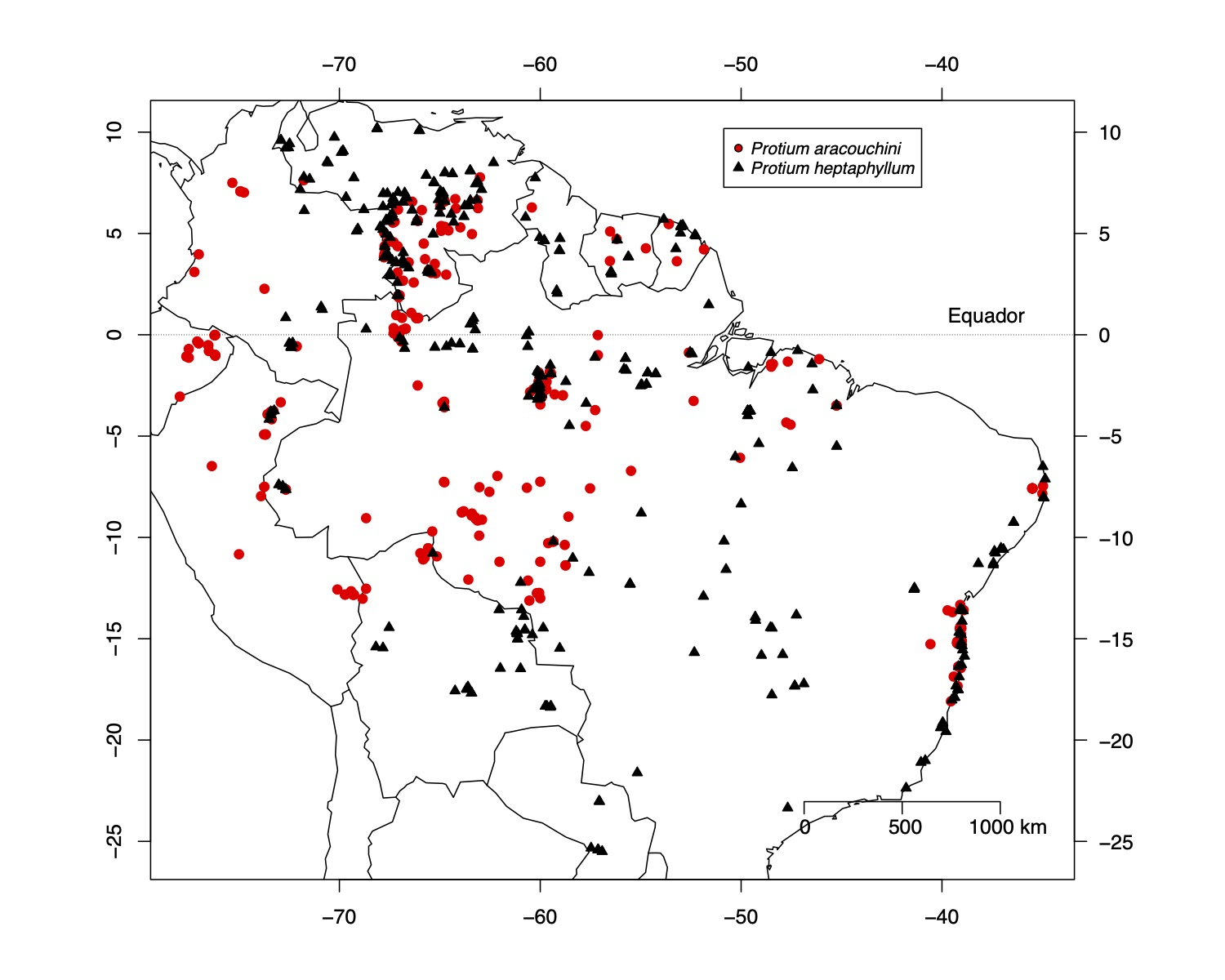
Figura 1: Distribuição geográfica de Protium aracouchini (Aubl.) Marchand e P. heptaphyllum (Aubl.) Marchand segundo dados do herbário do Jardim Botânico de Nova Iorque (NYBG). Dados já desatualizados, baixados em 2015 da base de dados do NYBG.
Salva plot em um pdf
Pode-se salvar também este mapa em um pdf, utilizando a função pdf(). Veja abaixo.
### Cria um pdf e Plota o mapa
pdf('meu_mapa_complexo_protium_aracouchini_e_heptaphyllum.pdf',height=8,width=10)
map(regions = paises, fill = F, xlim = x1, ylim = y1)
points(long,lat, pch = protium$pontos, col = cores.map, bg = cores.map, cex = protium$cex.p)
map.axes()
axis(side=4,las=1)
axis(side=3,las=1)
par(cex=1, las=1)
map.scale(max(long) - 12, ratio = F, cex = 1, metric = T)
abline(h=0,lwd=0.5,lty="dotted")
text(x = max(long) - 1, y = 1,labels="Equador", cex=1, adj=c(1,0.5))
legend(max(long) - 16 , max(lat),legend = spp, pch = unique(protium$pontos), pt.bg = unique(cores.map), cex = 0.8, x.intersp = 0.8, text.font = 3)
dev.off()
Informações sobre esta postagem
Esta postagem foi escrita em ambiente R utilizando os pacotes R Markdown e knitr.
Dúvidas, sugestões, erros no script, favor entrar em contato comigo, via comentários, ou por email.
Esta postagem completa, incluindo os blocos de código, podem ser lidos inteiramente neste link. Lá você pode copiar o arquivo bruto para o seu computador e modificá-lo à vontade.
Divirta-se!!!
Saiba mais
- Making maps with R
- Maps in R: Introduction
- R notebooks
- Why I love R Notebooks
- Advantages of Using R Notebooks For Data Analysis Instead of Jupyter Notebooks
- Reproducible Research Using RMarkdown and Git through RStudio
- Posted on:
- March 6, 2020
- Length:
- 7 minute read, 1331 words