Baixando dados de repositórios virtuais de dados
By Ricardo de Oliveira Perdiz in tutorial repositórios virtuais dados ecológicos dados taxonômicos
April 3, 2020
Postagem preparada para a disciplina BOT-89 Introdução ao R e Preparação de Dados, ligada ao Programa de Pós-graduação em Ciências Biológicas (Botânica) (PPGBOT) do Instituto Nacional de Pesquisas da Amazônia (INPA), Amazonas, Brasil, e ministrada anualmente pelo Dr. Alberto Vicentini (INPA), com minha participação este ano como professor.
Neste tutorial, aprenderemos a baixar dados de dois diferentes repositórios para utilizá-los durante o decorrer de nossa disciplina de aprendizado da linguagem R.
Dados a serem baixados
Vamos trabalhar com conjuntos de dados variados. Para baixá-los, podemos visitar cada link abaixo e baixar um a um, ou então utilizar o R para baixar diretamente no seu computador por meio de algumas funções oriundas de determinados pacotes. Abaixo segue a lista de conjuntos de dados:
-
Costa et al. (2020) - dados de ocorrências de espécies plantas de campinaranas em alguns locais na Amazônia - Baixe os dados neste link, buscando pelo botão
Download dataset: https://datadryad.org/stash/dataset/doi:10.5061/dryad.gmsbcc2hr. -
Jaramillo et al. (2019) - dados de diversidade de plantas em parcelas espalhadas por ilhas de mata ao redor de Boa Vista, Roraima, Brasil. Baixe os dados neste link, buscando pelo botão
Download: https://www.gbif.org/dataset/5846793d-f540-473c-88ee-23b2b13af7ef. -
Silva et al. (2019) - dados de diversidade de árvores em parcelas permanentes na Estação Ecológica de Maracá, Roraima, Brasil. Baixe os dados neste link, buscando pelo botão
Download: https://www.gbif.org/dataset/01d28467-87a1-4d64-ba40-4e3e1cc9091b.
Baixando dados manualmente
Caso queira baixar os dados manualmente, darei um exemplo de como baixar os dados de Costa et al. (2020), depositado no Dryad, e os dados de Jaramillo et al. (2019), depositados no Sibbr/GBIF.
Dados de Costa et al. (2020)
Os dados podem ser baixados em https://datadryad.org/stash/dataset/doi:10.5061/dryad.gmsbcc2hr.

Busque no canto direito o botão Download dataset. Ao apertá-lo, automaticamente haverá a opção de salvar o arquivo (ou dependendo das configurações de seu computador, o arquivo será automaticamente salvo, por padrão, na pasta Download).
Por padrão, os dados vêm zipados (se você não sabe o que é um arquivo zipado,
veja aqui o que é).
Para tirar o zip, basta clicar no arquivo duas vezes que uma pasta se abrirá (ou haverá a opção de escolher o local onde se devem armazenar os arquivos contidos dentro do arquivo .zip; caso isso não ocorra, entre em contato comigo para desvendar o que aconteceu).
Dentro deste arquivo .zip, há apenas dois arquivos: um chamado Costa_etal_data.RData, e um metadado, metadata.txt.
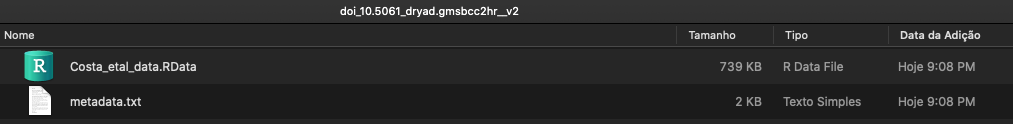
O arquivo .RData é próprio do
R e pode conter um ou mais objetos.
Você pode aprender mais sobre como manipular este tipo de arquivo dentro do R lendo o
material de nosso curso da linguagem, disponível gratuitamente no endereço
https://intror.netlify.app.
Dados de Jaramillo et al. (2019)
Os dados podem ser baixados em
https://www.gbif.org/dataset/5846793d-f540-473c-88ee-23b2b13af7ef.
Busque o botão Download abaixo do título.
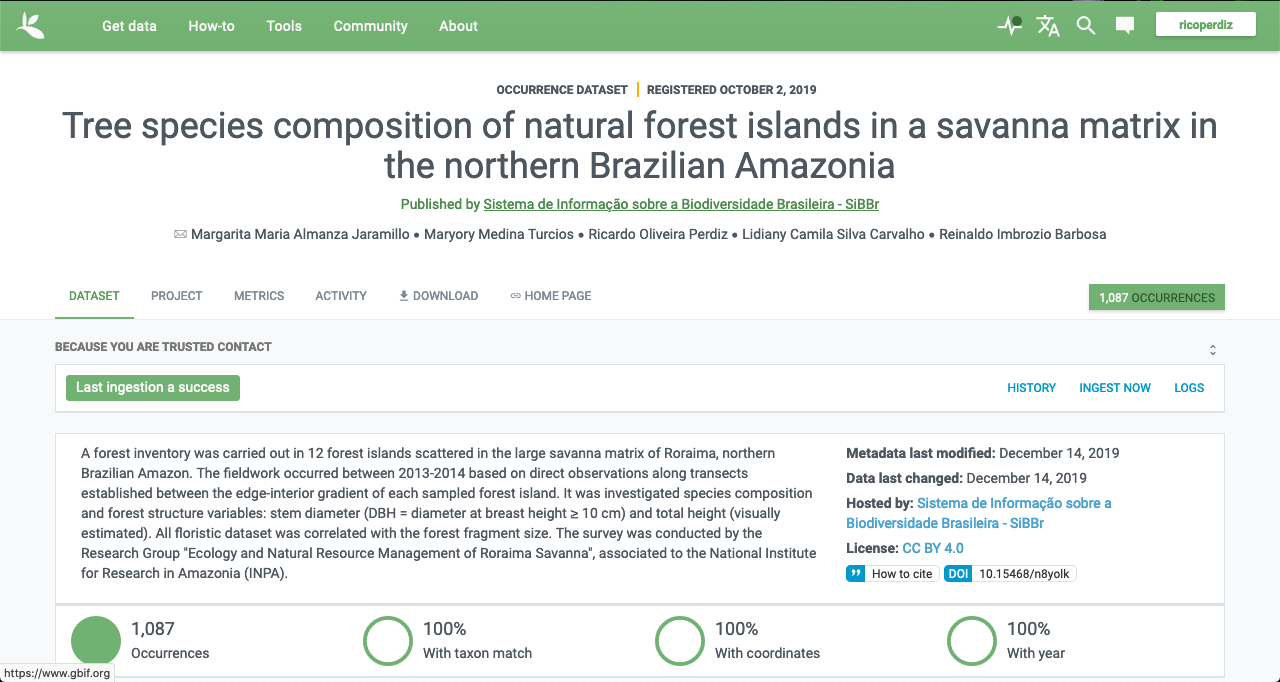
Após pressionar o botão Download, uma caixa se abre com três opções:
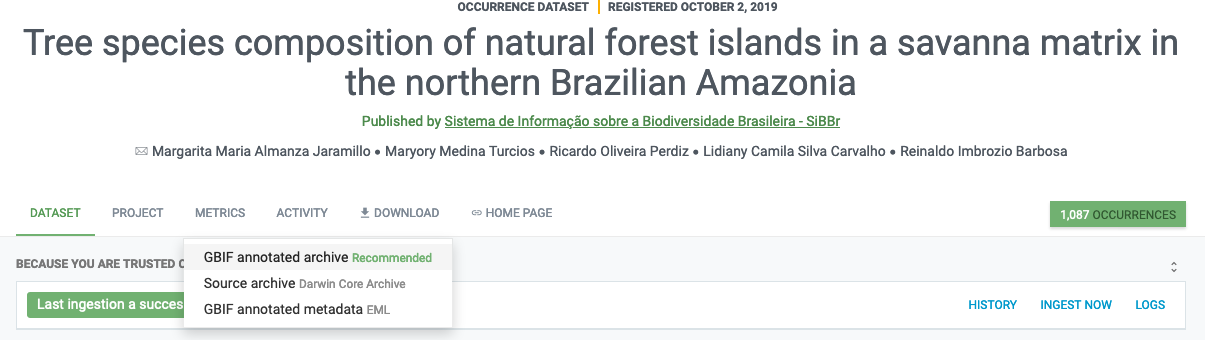
* GBIF annotated Archive `Recommended`
* Source archive `Darwin Core Archive`
* GBIF annotated metadata `EML`
Eu escolhi baixar no formato Darwin Core Archive. Porém, vamos selecionar a opção GBIF annotated archive. Isso nos levará a outra página, em que todas as opções para baixar serão expostas, e alguns avisos importantes aparecerão, durante o processo:
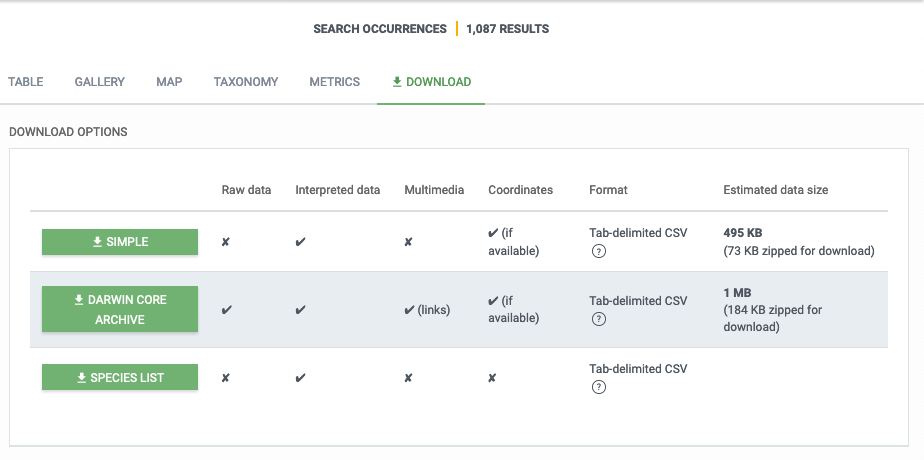
Selecionando a opção do meio, recebemos um aviso:
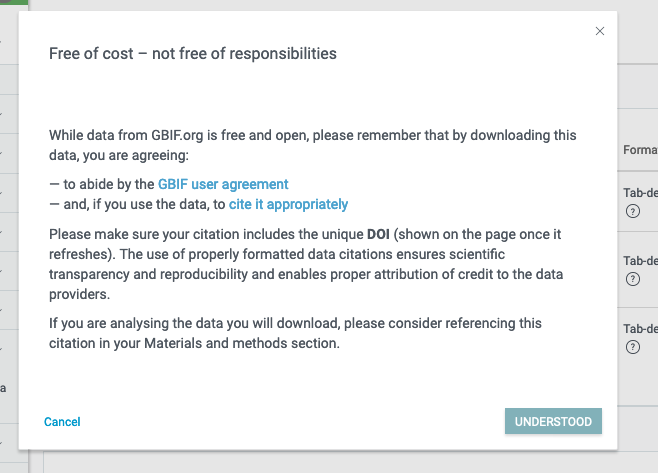
Ao confirmarmos a opção Understood, isso nos leva à página de preparo do download:
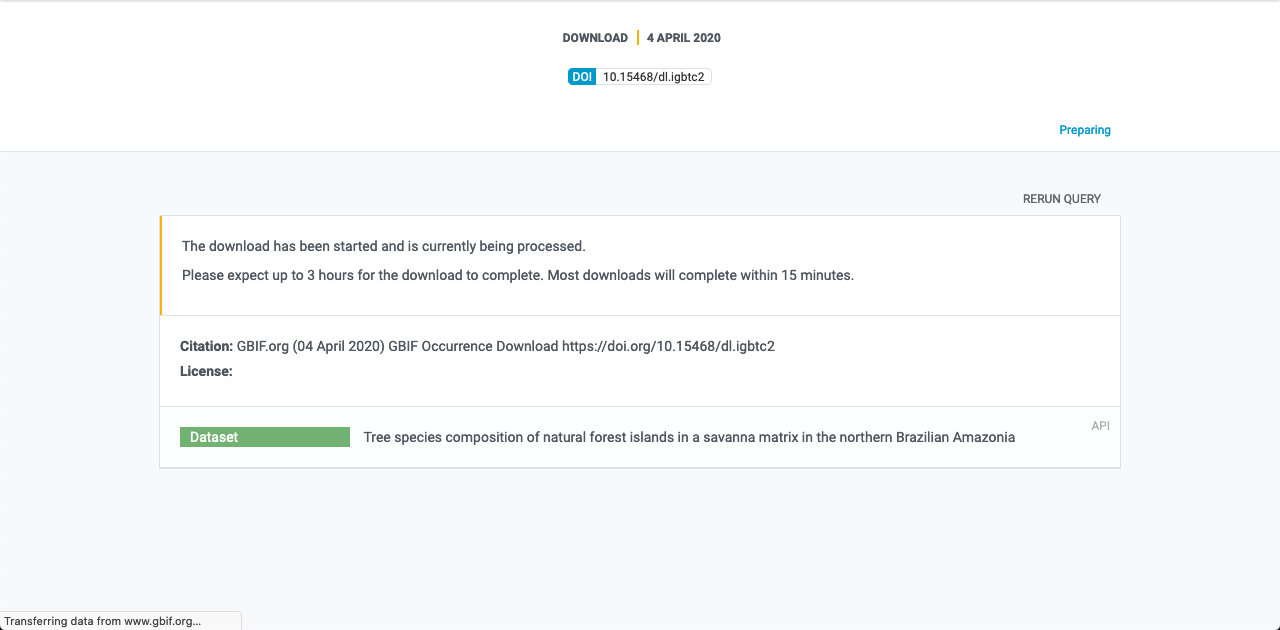
Ao terminar o preparo, nesta mesma página aparecerá a opção de baixar os dados e também será enviado ao seu email um link para que possas baixar o conjunto de dados.
Assim como os dados baixados do Dryad, os dados também vêm zipados. Faça o mesmo procedimento adotado para os dados do Dryad, e veja o que existe dentro do .zip:
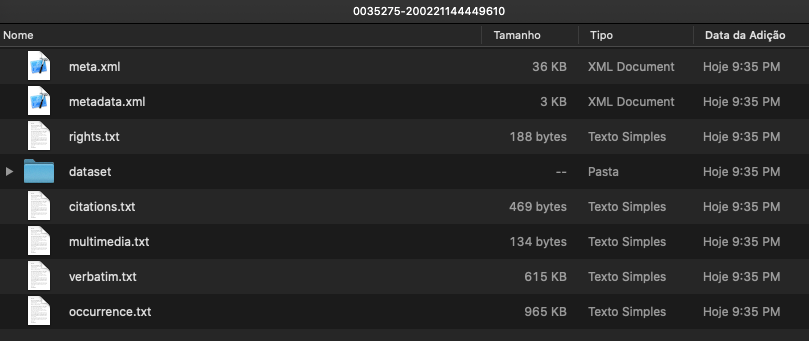
Os dados estão dentro do arquivo occurrence.txt. Você pode guardar o conjunto inteiro, preferencialmente, ou apenas este arquivo occurrence.txt. Sugiro que guarde a pasta inteira para uso nos próximos passos da disciplina.
Esse procedimento, apesar de diferir em detalhes de repositório para repositório, é bem semelhante em todos os repositórios e não é nada complexo. Caso haja dúvidas, não hesite em me contatar.
Baixando os dados usando o R
Por enquanto, apenas o pacote que permite baixar dados GBIF (Chamberlain, Oldoni, e Waller 2022) está funcionando plenamente. O pacote que baixa dados do DRYAD está sendo reparado no momento devido à mudanças no API do repositório ( veja aqui). Por esta razão, vamos focar em baixar os dados estocados no GBIF por enquanto. Primeiro passo é baixar o pacote, caso você não o tenha instalado em seu computador:
install.packages("rgbif")
Após instalá-lo, não esqueça de importá-lo à sessão de trabalho:
library("rgbif")
Dados no GBIF
Para baixar dados no GBIF utilizando o rgbif, podemos buscar dados de ocorrência de espécies, amostras em localidades específicas, ou conjuntos de dados, utilizando as funções de busca que o pacote fornece, em especial a função occ_search().
Demonstraremos abaixo o uso básico de como executar algumas dessas ações.
Baixando dados por meio de buscas
Podemos buscar ocorrências de uma determinada espécie:
occ_search(scientificName='Protium aracouchini', limit = 10)
## Records found [3046]
## Records returned [10]
## No. unique hierarchies [1]
## No. media records [10]
## No. facets [0]
## Args [limit=10, offset=0, scientificName=Protium aracouchini, fields=all]
## # A tibble: 10 × 82
## key scientificName decimalLatitude decimalLongitude issues datasetKey
## <chr> <chr> <dbl> <dbl> <chr> <chr>
## 1 3321913475 Protium aracou… 3.53 -73.4 "" 1035299a-…
## 2 3321914313 Protium aracou… 3.53 -73.4 "" 1035299a-…
## 3 3321914314 Protium aracou… 3.53 -73.4 "" 1035299a-…
## 4 3321914315 Protium aracou… 3.53 -73.4 "" 1035299a-…
## 5 3321914316 Protium aracou… 3.53 -73.4 "" 1035299a-…
## 6 3321914317 Protium aracou… 3.53 -73.4 "" 1035299a-…
## 7 3321914318 Protium aracou… 3.53 -73.4 "" 1035299a-…
## 8 3321914319 Protium aracou… 3.53 -73.4 "" 1035299a-…
## 9 3321914320 Protium aracou… 3.53 -73.4 "" 1035299a-…
## 10 3321914321 Protium aracou… 3.53 -73.4 "" 1035299a-…
## # … with 76 more variables: publishingOrgKey <chr>, installationKey <chr>,
## # publishingCountry <chr>, protocol <chr>, lastCrawled <chr>,
## # lastParsed <chr>, crawlId <int>, projectId <chr>, programmeAcronym <chr>,
## # hostingOrganizationKey <chr>, basisOfRecord <chr>, occurrenceStatus <chr>,
## # taxonKey <int>, kingdomKey <int>, phylumKey <int>, classKey <int>,
## # orderKey <int>, familyKey <int>, genusKey <int>, speciesKey <int>,
## # acceptedTaxonKey <int>, acceptedScientificName <chr>, kingdom <chr>, …
Ou um de determinado gênero:
occ_search(scientificName = 'Protium', limit = 10)
## Records found [65331]
## Records returned [10]
## No. unique hierarchies [6]
## No. media records [10]
## No. facets [0]
## Args [limit=10, offset=0, scientificName=Protium, fields=all]
## # A tibble: 10 × 106
## key scientificName decimalLatitude decimalLongitude issues datasetKey
## <chr> <chr> <dbl> <dbl> <chr> <chr>
## 1 3044641864 Protium aguila… 8.68 -83.7 "cdro… 50c9509d-…
## 2 3044893837 Protium santam… 8.68 -83.8 "cdro… 50c9509d-…
## 3 3047723316 Protium ovatum… NA NA "colm… b3769e61-…
## 4 3313936315 Protium Burm.f… -20.6 -40.4 "cdro… 4300f8d5-…
## 5 3397855715 Protium spruce… -10.7 -47.0 "cudc… e38d4c95-…
## 6 3109019507 Protium santam… 8.78 -83.4 "cdro… 50c9509d-…
## 7 3321913301 Protium glabre… 3.52 -73.4 "" 1035299a-…
## 8 3321913302 Protium glabre… 3.53 -73.4 "" 1035299a-…
## 9 3321913303 Protium glabre… 3.52 -73.4 "" 1035299a-…
## 10 3321913304 Protium glabre… 3.53 -73.4 "" 1035299a-…
## # … with 100 more variables: publishingOrgKey <chr>, installationKey <chr>,
## # publishingCountry <chr>, protocol <chr>, lastCrawled <chr>,
## # lastParsed <chr>, crawlId <int>, hostingOrganizationKey <chr>,
## # basisOfRecord <chr>, occurrenceStatus <chr>, taxonKey <int>,
## # kingdomKey <int>, phylumKey <int>, classKey <int>, orderKey <int>,
## # familyKey <int>, genusKey <int>, speciesKey <int>, acceptedTaxonKey <int>,
## # acceptedScientificName <chr>, kingdom <chr>, phylum <chr>, order <chr>, …
Podemos buscar dados de ocorrências por estados, países, localidades.
Isso pode ser feito para um ou mais locais.
Primeiro, devemos buscar a chave referente às plantas vasculares.
Para isso, utilizaremos a função name_suggest:
pvas <- name_suggest(q = "Tracheophyta")
pvas
## Records returned [1]
## No. unique hierarchies [0]
## Args [q=Tracheophyta, limit=100, fields1=key, fields2=canonicalName,
## fields3=rank]
## # A tibble: 1 × 3
## key canonicalName rank
## <int> <chr> <chr>
## 1 7707728 Tracheophyta PHYLUM
Agora, com essa chave, podemos restringir nossas buscas à localidade desejada somente para o táxon de interesse, que são as plantas vasculares.
Podemos fazer isso através de uma chave presente no objeto pvas, criado acima, e que pode ser checada com o comando pvas$key.
Para buscar os dados de localidade, vamos restringir nossas buscas ao estado do Amazonas, Brasil:
Vamos agora realizar a busca para dois estados. Vamos incluir Rondônia na busca:
occ_search(
stateProvince = c("Amazonas","Rondônia"),
phylumKey = pvas$key,
limit = 50,
return = "data" )
Devemos atentar que por padrão os resultados de operações com occ_search são limitados a 500 registros.
Demonstramos aqui apenas o uso básico dessas operações.
Sugiro que visitem os tutoriais do pacote rgbif disponíveis nas páginas abaixo para entenderem melhor as diversas funcionalidades que ele oferece:
- Página GitHub do pacote rgbif;
- Introduction to rgbif;
- Downloading data from GBIF;
- Taxonomic names;
- Cleaning data using GBIF issues.
Baixando por meio de uma chave
Se desejamos baixar um conjunto de dados específico, nós devemos buscar a chave GBIF deste conjunto de dados na página GBIF do mesmo.
Para obter essa chave, é necessário que visitemos a página GBIF que contem os dados desejados para copiarmos o endereço da página.
Após copiar este endereço, devemos retirar do endereço o seguinte trecho: https://www.gbif.org/dataset/. O que sobrar, é a chave GBIF de nosso interesse.
Vamos demonstrar esta rotina de trabalho com dois conjuntos de dados: Silva et al. (2019) e Jaramillo et al. (2019).
Silva et al. 2019
Vamos começar a baixar os dados de Silva et al. (2019). O endereço para baixar estes dados está exposto abaixo:
https://www.gbif.org/dataset/01d28467-87a1-4d64-ba40-4e3e1cc9091b
Retirando o trecho https://www.gbif.org/dataset/, temos então a chave GBIF de interesse:
01d28467-87a1-4d64-ba40-4e3e1cc9091b
Antes de partir para a função occ_download, devemos utilizar a família de funções pred para estabelecer o filtro de dados que serão baixados do GBIF.
Essa família de funções consiste basicamente de dois argumentos: key e value.
O argumento key aceita o nome das colunas GBIF; por exemplo, se desejamos realizar uma busca nos estados, utilizamos o termo “stateProvince” em key (veja mais detalhes na ajuda da função teclando ?pred); se desejamos baixar um conjunto de dados específico, utilizamos o termo “datasetKey” em key. Já o argumento value é onde devemos digitar os valores referentes à chave (key).
Por fim, devemos atentar a três argumentos indispensáveis da função occ_download: user, seu nome de usuário na plataforma; pwd, sua senha de entrada no sistema, e email, seu email utilizado no seu cadastro.
Sugiro que você os associe a cada um dos objetos abaixo. Substitua os textos pelos valores verdadeiros.
Ao atribuí-los, esses valores estarão disponíveis para utilizarmos na função occ_download.
gbif_user <- "ColoqueAquiSeuNomeDeUsuario"
gbif_pwd <- "ColoqueAquiSuaSenha"
gbif_email <- "ColoqueAqui@SeuEmail"
Uma vez estabelecidos os valores de usuário, senha, email, e também os filtros do download, podemos agora utilizar a função occ_download:
res1 <-
occ_download(
pred("datasetKey", value = "01d28467-87a1-4d64-ba40-4e3e1cc9091b"),
user = gbif_user,
pwd = gbif_pwd,
email = gbif_email
)
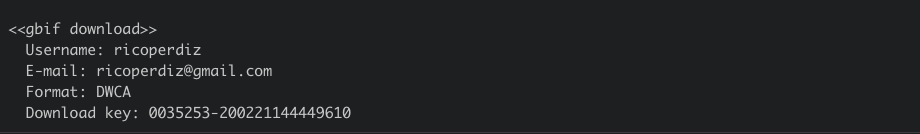
O resultado desta ação gera uma chave: Download key: 0035253-200221144449610. Para você, leitor, o resultado será outra chave, então preste atenção e não se confunda.
Peguemos então esta chave e passemos para a próxima função, occ_download_meta, que checará o status do download. O status pode ser visto na variável Status, que aparece após se executar occ_download_meta(res1), que possui a variável res1, obtida quando rodamos o comando acima.
occ_download_meta(res1)
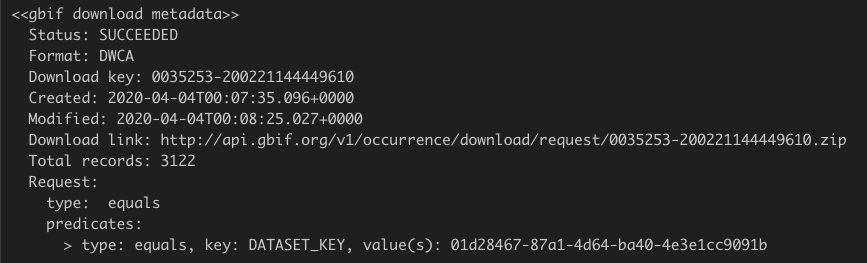
Devemos rodar várias vezes o comando abaixo até que o status do download mude para SUCCEEDED.
Ao se obter sucesso no download, prossigamos para o próximo passo, que é o de baixar os dados propriamente ditos, utilizando a função occ_download_get.
silvaetal2019 <- occ_download_get(res1, path = ".")
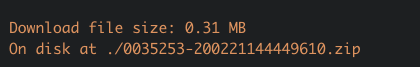
A função occ_download_get apenas baixa os dados para o seu computador dentro da sua pasta de trabalho.
Esta função não importa os dados para o R.
Para fazer isso, usemos a função occ_download_import, que transforma os dados em um data.frame.
silvaetal2019_df <- occ_download_import(silvaetal2019)
silvaetal2019_df
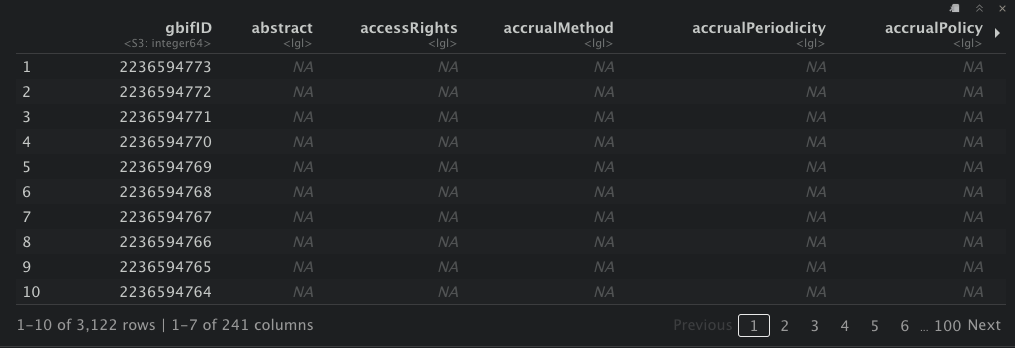
Agora você pode salvar esse data.framecomo um arquivo .csv dentro da sua pasta de trabalho ou onde você desejar.
write.table(silvaetal2019_df, file = "silvaetal2019.csv", sep = '\t', quote = TRUE)
Jaramillo et al. 2019
Vamos agora baixar os dados de Jaramillo et al. (2019).
A chave desse conjunto de dados é 5846793d-f540-473c-88ee-23b2b13af7ef. Vamos seguir o mesmo procedimento adotado acima.
res2 <-
occ_download(
pred("datasetKey", value = "5846793d-f540-473c-88ee-23b2b13af7ef"),
user = gbif_user,
pwd = gbif_pwd,
email = gbif_email
)
occ_download_meta(res2)
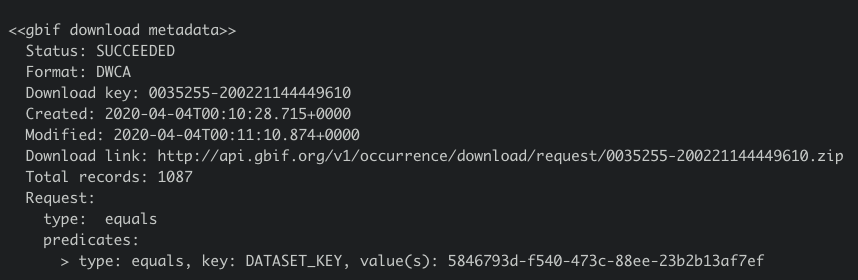
jaramilloetal2019 <- occ_download_get(res2, path = ".")
jaramilloetal2019_df <- occ_download_import(jaramilloetal2019)
jaramilloetal2019_df
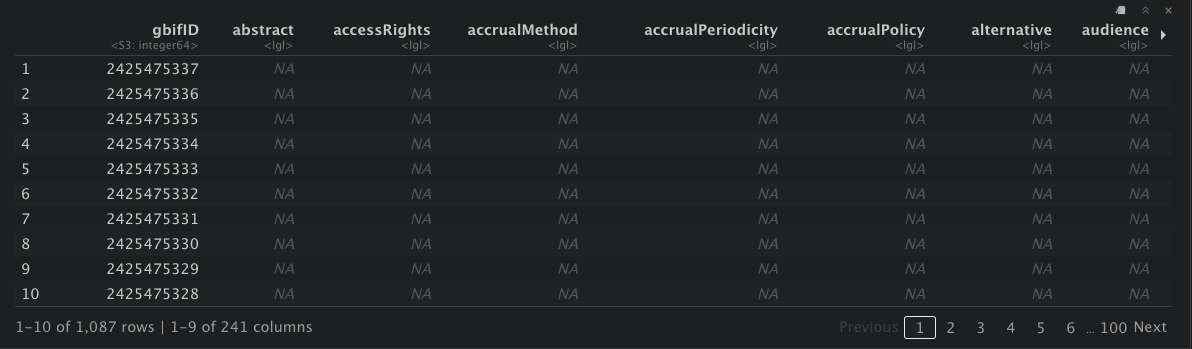
write.table(jaramilloetal2019_df, file = "jaramilloetal2019.csv", sep = '\t', quote = TRUE)
Sempre que utilizar um conjunto de dados em uma publicação, cite o conjunto de dados! Como exemplo, veja abaixo como obter a citação de um conjunto de dados depositado no GBIF.
Citação do conjunto de dados
Embora não seja um artigo científico, conjuntos de dados depositados em repositórios de dados possuem um DOI, portanto, o conjunto de dados deve ser citado caso seja utilizado, conforme instrução do próprio GBIF.
Para obter uma citação de um conjunto de dados do GBIF, por exemplo, o pacote rgbif fornece a função gbif_citation, que deve ser utilizada em conjunto com o objeto resultante da função occ_download_import. Tomando os dados de Silva et al. (2019) como exemplo, temos:
gbif_citation(silvaetal2019)
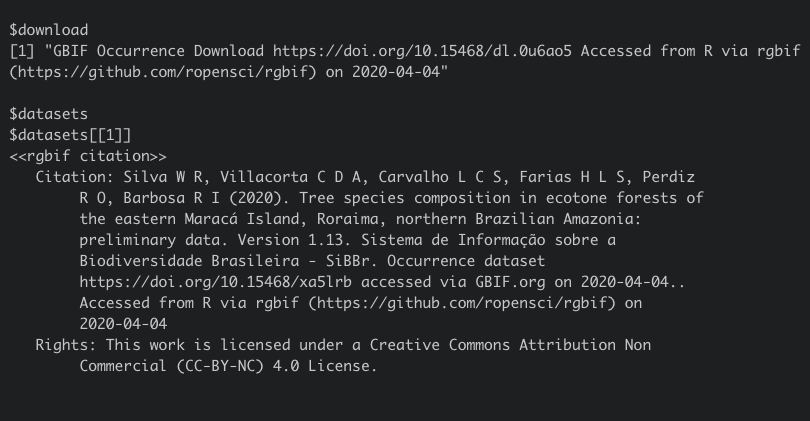
Repare que há dois objetos resultantes, um chamado download, e outro chamado dataset, ambos apresentando um DOI. O primeiro se refere ao conjunto de dados baixado via
rgbif ; caso você baixe
manualmente, você pode também encontrar um DOI para esse download manual. O segundo se refere ao DOI do conjunto de dados propriamente dito e armazenado no repositório de dados.
Aqui termina este breve tutorial para baixar dados de repositórios virtuais.
Por enquanto, utilizamos apenas um pacote do R, rgbif, para baixar dados diretamente do GBIF utilizando o ambiente R.
Em breve, atualizarei esta postagem com outros pacotes, próprios para baixar de outros repositórios.
Qualquer dúvida, sugestão, deixe um comentário ou entre em contato comigo via Github, Twitter, email, o que você achar melhor.
Até a próxima!
Referências
Chamberlain, Scott, Damiano Oldoni, e John Waller. 2022. rgbif: Interface to the Global Biodiversity Information Facility API. https://CRAN.R-project.org/package=rgbif.
Costa, Flavio M., Mario H. Terra-Araujo, Charles E. Zartman, Cintia Cornelius, Fernanda A. Carvalho, Michael J. G. Hopkins, Pedro L. Viana, Eduardo M. B. Prata, e Alberto Vicentini. 2020. “Islands in a green ocean: Spatially structured endemism in Amazonian white-sand vegetation”. Biotropica 52 (1): 34–45. https://doi.org/10.1111/btp.12732.
Jaramillo, M. M. A., M. M. Turcios, R. O. Perdiz, L. C. S. Carvalho, e R. I. Barbosa. 2019. “Tree species composition of natural forest islands in a savanna matrix in the northern Brazilian Amazonia. v1.9”. Sistema de Informação sobre a Biodiversidade Brasileira - SiBBr. https://doi.org/10.15468/n8yolk.
Silva, W. R., C. D. A. Villacorta, L. C. S. Carvalho, H. L. S. Farias, R. O. Perdiz, e R. I. Barbosa. 2019. “Tree species composition in ecotone forests on Maracá Island, Roraima, northern Brazilian Amazonia: preliminary data. V1.18”. Sistema de Informação sobre a Biodiversidade Brasileira - SiBBr. https://doi.org/10.15468/xa5lrb.
- Posted on:
- April 3, 2020
- Length:
- 12 minute read, 2453 words